mmseg教程
摘要
主要是记录如何使用mmseg这个分割库,使用自己的数据集进行训练,使用自己的config参数以及最后的测试以及推理。
在这里主要记录的是使用Unet网络的应用,数据采用胃部肠化数据,共有两个标签,一个是正常组织的标签(蓝色),一个是肠化组织标签(红色)。
我们的任务就是训练一个网络来识别正常腺体和异常腺体的区域。
mmseg介绍
就是一个分割工具箱没什么好介绍的,具体内容看链接:
- 欢迎来到 MMSegmentation 的文档! — MMSegmentation 1.2.1 文档
- open-mmlab/mmsegmentation: OpenMMLab Semantic Segmentation Toolbox and Benchmark. (github.com)
内容具体介绍
- 环境配置
- 数据集制作
- 自定义数据集
- 数据集的读取
- 模型选取以及参数调整
- 训练测试
主要参考来源:mmsegmentation教程1:自定义数据集、config文件修改、训练教程_AESA相控阵的博客-CSDN博客
环境配置
跟着官方的来就行开始:安装和运行 MMSeg — MMSegmentation 1.2.1 文档
可能会遇到问题,遇到问题装一个这个
pip install mmcv-full数据集制作
首先自己的数据要按照如下的格式摆放:
├── data
│ ├── my_dataset
│ │ ├── img_dir
│ │ │ ├── train
│ │ │ │ ├── xxx{img_suffix}
│ │ │ │ ├── yyy{img_suffix}
│ │ │ │ ├── zzz{img_suffix}
│ │ │ ├── val
│ │ ├── ann_dir
│ │ │ ├── train
│ │ │ │ ├── xxx{seg_map_suffix}
│ │ │ │ ├── yyy{seg_map_suffix}
│ │ │ │ ├── zzz{seg_map_suffix}
│ │ │ ├── val几个主要的要点:
- 这个data文件夹最好放在mmseg的子路径下,加快读取速度
- img_dir放置所有图片,ann_dir放置所有mask(标注文件),提前分好测试集和训练集(分训练集和测试集代码如下),注意文件名要对应(img中在测试集的xxx文件,它的标注文件一定也在测试集中)
import os import random import shutil random.seed(42) input_img_dir = '' input_label_dir = '' output_img_dir = '' output_label_dir = '' for split in ['train', 'val']: os.makedirs(os.path.join(output_img_dir, split), exist_ok=True) os.makedirs(os.path.join(output_label_dir, split), exist_ok=True) img_files = os.listdir(input_img_dir) label_files = os.listdir(input_label_dir) train_ratio = 0.7 val_ratio = 0.3 random.shuffle(img_files) total_samples = len(img_files) num_train = int(total_samples * train_ratio) train_img_files = img_files[:num_train] val_img_files = img_files[num_train:] for src_dir, split in [(input_img_dir, 'train'), (input_img_dir, 'val')]: for img_file in locals()[f'{split}_img_files']: label_file = img_file # 图像和标签文件名一一对应 src_img_path = os.path.join(input_img_dir, img_file) src_label_path = os.path.join(input_label_dir, label_file) dst_img_path = os.path.join(output_img_dir, split, img_file) dst_label_path = os.path.join(output_label_dir, split, label_file) shutil.move(src_img_path, dst_img_path) shutil.move(src_label_path, dst_label_path)
自定义数据集
由于我们的任务比较特殊,常规的分割方法所采用的数据集,因此需要自己重新定义。
在mmseg/datasets/目录下找到stare.py文件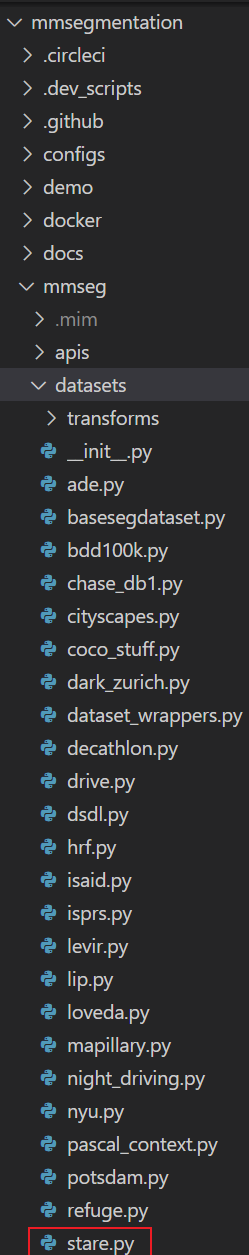
同路径下复制一份,重命名为my_dataset.py记住这个名字后续还要用。
下面需要修改三个地方: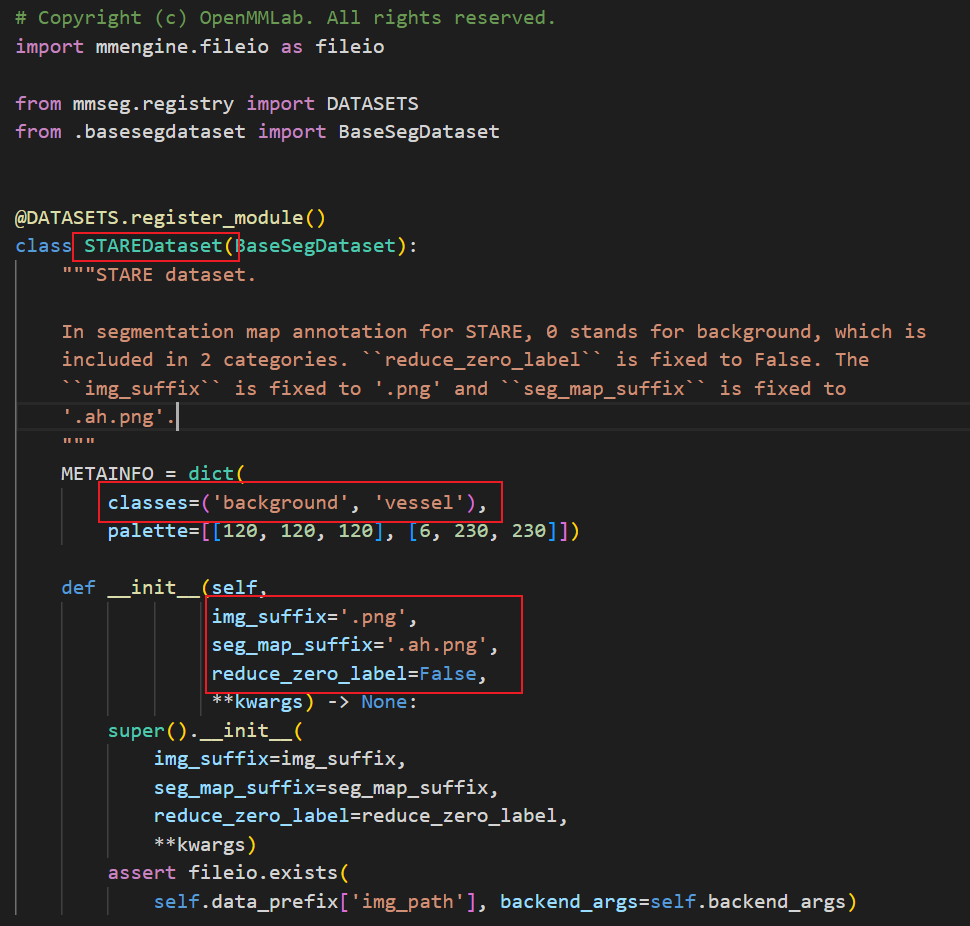
你需要修改标红的三个地方:
STAREDataset:数据集的名称,自定义,自己要记得
CLASSES:表示你数据集的背景+类别
PALETTE:表示你数据集各类别的像素值
img_suffix:原图图像后缀
seg_map_suffix:mask图像后缀
reduce_zero_label:这个后面说,算了现在说吧。这个就是是否去除背景,我们做的是实例分割不是语义分割,是可以去除背景的,这里把背景去掉,实际上准确率会高很多。写给 MMSegmentation 工具箱新手的避坑指南 - 知乎 (zhihu.com)
# Copyright (c) OpenMMLab. All rights reserved.
import mmengine.fileio as fileio
from mmseg.registry import DATASETS
from .basesegdataset import BaseSegDataset
@DATASETS.register_module()
class Mydataset(BaseSegDataset):
"""STARE dataset.
In segmentation map annotation for STARE, 0 stands for background, which is
included in 2 categories. ``reduce_zero_label`` is fixed to False. The
``img_suffix`` is fixed to '.png' and ``seg_map_suffix`` is fixed to
'.ah.png'.
"""
METAINFO = dict(
classes=('blue', 'red'),
palette=[[120, 120, 120], [6, 230, 230]])
def __init__(self,
img_suffix='.png',
seg_map_suffix='.png',
reduce_zero_label=True,
**kwargs) -> None:
super().__init__(
img_suffix=img_suffix,
seg_map_suffix=seg_map_suffix,
reduce_zero_label=reduce_zero_label,
**kwargs)
assert fileio.exists(
self.data_prefix['img_path'], backend_args=self.backend_args)最后修改后的样子
然后修改这个__init__文件
前面添加一个from .my_dataset import Mydataset
后面添加一个, ‘Mydataset’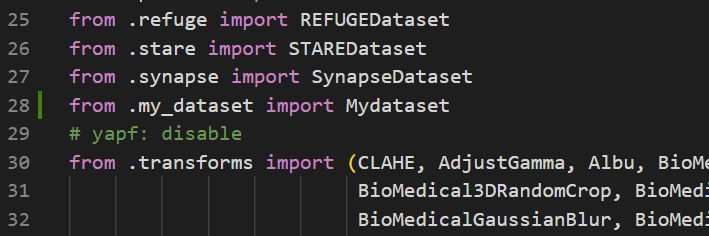
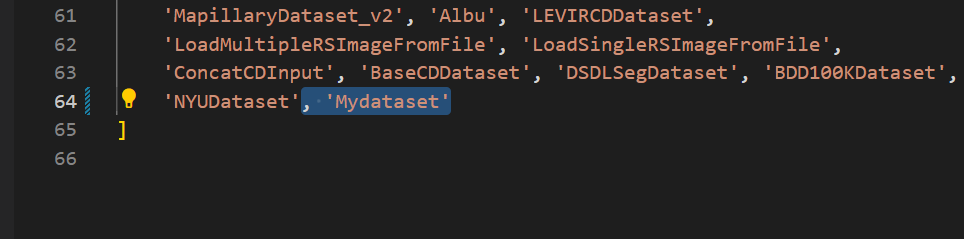
就是你刚刚命名的文件名和dataset的命名
注意:palette是调色板,对应与后续测试的时候每类别的输出颜色,另外,mask也有要求,由于现在的标签是蓝色和红色,即[255, 0, 0]和[0, 0, 255]。但是后续的处理需要变为0:背景,1:标签1,2:标签2。因此标签需要进一步处理。
代码如下:
import os
from tqdm import tqdm
import numpy as np
from PIL import Image
input_folder = ''
output_folder = ''
for filename in tqdm(os.listdir(input_folder)):
if filename.endswith(".png"):
img_path = os.path.join(input_folder, filename)
img = Image.open(img_path)
img_array = np.array(img)
img_array[np.where((img_array == [0, 0, 255]).all(axis=2))] = [1, 1, 1]
img_array[np.where((img_array == [255, 0, 0]).all(axis=2))] = [2, 2, 2]
mask = img_array[:, :, 0]
new_img = Image.fromarray(mask)
new_img.save(os.path.join(output_folder, filename))数据集的读取
在configs/_ base_ /datasets/目录下找到stare.py文件,复制一份重命名为my_dataset.py。进行修改
修改后的代码以及所需要修改的部分都已经添加注释了
# dataset settings
dataset_type = 'MyDataset' # 改成自己的数据集类名
data_root = '/data_sda/data/gastric_2label' # 数据集存储路径
img_scale = (256, 256) # 图像尺寸
crop_size = (256, 256) # 裁剪尺寸
train_pipeline = [
dict(type='LoadImageFromFile'),
dict(type='LoadAnnotations'),
dict(type='Resize', scale=img_scale, keep_ratio=True), #这个改一下
dict(type='RandomCrop', crop_size=crop_size, cat_max_ratio=0.75),
dict(type='RandomFlip', prob=0.5),
dict(type='PhotoMetricDistortion'),
dict(type='PackSegInputs')
]
test_pipeline = [
dict(type='LoadImageFromFile'),
dict(type='Resize', scale=img_scale, keep_ratio=True),
# add loading annotation after ``Resize`` because ground truth
# does not need to do resize data transform
dict(type='LoadAnnotations'),
dict(type='PackSegInputs')
]
img_ratios = [0.5, 0.75, 1.0, 1.25, 1.5, 1.75]
tta_pipeline = [
dict(type='LoadImageFromFile', backend_args=None),
dict(
type='TestTimeAug',
transforms=[
[
dict(type='Resize', scale_factor=r, keep_ratio=True)
for r in img_ratios
],
[
dict(type='RandomFlip', prob=0., direction='horizontal'),
dict(type='RandomFlip', prob=1., direction='horizontal')
], [dict(type='LoadAnnotations')], [dict(type='PackSegInputs')]
])
]
train_dataloader = dict(
batch_size=12, # 这个可以自己测试
num_workers=1, #
persistent_workers=True,
sampler=dict(type='InfiniteSampler', shuffle=True),
dataset=dict(
type='RepeatDataset',
times=40000,
dataset=dict(
type=dataset_type,
data_root=data_root,
data_prefix=dict(
img_path='img_split/train', # 改路径
seg_map_path='label_split/train'), # 改路径
pipeline=train_pipeline)))
val_dataloader = dict(
batch_size=1,
num_workers=4,
persistent_workers=True,
sampler=dict(type='DefaultSampler', shuffle=False),
dataset=dict(
type=dataset_type,
data_root=data_root,
data_prefix=dict(
img_path='img_split/val', # 改路径
seg_map_path='label_split/val'), # 改路径
pipeline=test_pipeline))
test_dataloader = val_dataloader
val_evaluator = dict(type='IoUMetric', iou_metrics=['mDice', 'mIoU', 'mFscore']) # 改一下评价指标
test_evaluator = val_evaluator
模型选取以及参数调整
这里简单写一个用Unet的(其他的其实类似)
比如找个这个./configs/unet/unet-s5-d16_deeplabv3_4xb4-40k_hrf-256x256.py
然后修改成这个
_base_ = [
'../_base_/models/deeplabv3_unet_s5-d16.py', '../_base_/datasets/my_dataset.py',
'../_base_/default_runtime.py', '../_base_/schedules/schedule_20k.py'
]
crop_size = (256, 256)
data_preprocessor = dict(size=crop_size)
model = dict(
data_preprocessor=data_preprocessor,
test_cfg=dict(crop_size=(256, 256), stride=(170, 170)))deeplabv3_unet_s5-d16:主要调用的网络架构,这里用的是Unet的架构
my_dataset:之前定义的那个数据集
default_runtime:基本架构,高级操作才会改一改
schedule_20k:训练策略,比如训练多少轮,按照iter训练还是epoch训练。
训练与测试
其实已经可以跑了,在模型里面有个num_classes参数,由于Unet本来就是2所以就不用调了。
python tools/train.py ./configs/unet/unet-20231113-mine.py --work-dir ./work-dir/test20231113直接开始愉快的训练,这样都跑不出来就自己对应看看什么问题吧。遇事不决可以先把mmseg删了再重装(可以解决小部分问题,比如在同一环境装了两个mmseg冲突了鼠鼠还真试过)
- 测试
测试我就不说这么多了,你都看到这了难道还不会找吗(其实是我还没写)